2012年,Douglas课题组在国际期刊Methods in Ecology and Evolution(MEE)发表了第一篇描述“生物多样性汤(Biodiversity Soup)”这一高通量条形码技术流程的文章,该文章已成为MEE期刊下载次数最高的文章之一。如今高通量条形码技术在环境保护和管理相关决策研究中的适用性已获得广泛的共识,但是若要在更广的领域应用该方法,还需要进一步减少PCR扩增、文库构建以及测序所带来的错误和偏好。
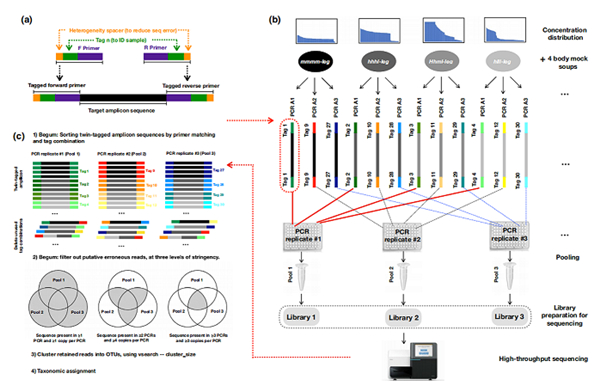
近日,该课题组在前期研究的基础上,进一步改进了高通量条形码技术流程,提高了运算速度,并能减少结果中的假阳性(如标签错配、嵌合体、错误序列)和假阴性。新流程改进了高通量条形码的实验设计和生物信息学分析,使用双胞胎标记法双向标记引物,将每个样本都进行多次独立的PCR扩增,并且通过qPCR来优化最终使用的退火温度和循环数。在生物信息学分析部分,采用的是Begum,它在样本拆分时可以忽略为了测序中平衡碱基而添加在标签上的几个碱基,并允许引物序列的错配且提高了运算速率。Begum能去除由于标签跳动所产生的假阳性序列,以及通过设置多个PCR重复和某一序列的重复出现次数来过滤PCR和测序等产生的错误序列。
该研究成果以“Biodiversity Soup II: A bulk-sample metabarcoding pipeline emphasizing error reduction”为题发表在国际期刊Methods in Ecology and Evolution上(文章链接:https://besjournals.onlinelibrary.wiley.com/doi/10.1111/2041-210X.13602)。中国科学院昆明动物研究所杨春燕为该文章的第一作者,Douglas Yu研究员为通讯作者。为了让更多人能学习、验证并设计适合自己的高通量条形码流程,文章还提供了研究中用于构建生物多样性汤所有物种的序列、Illumina测序数据、完整的分析命令脚本,以及用于引物标签设计的表格和指南。
该研究得到了遗传资源与进化国家重点实验室、动物进化与遗传前沿交叉卓越创新中心、国家自然科学基金委项目、中国科学院A类先导专项等机构和项目的资助。