| NATL SCI REV|发布全基因组单核苷酸变异数据库、涵盖近千个现存和古人族群 |
| 2019-10-31 | 作者: | 来源: 【打印】 |
2019年10月23日《国家科学评论》杂志在线发布了第一个藏族人群高质量参考基因组。该成果是动物进化与遗传前沿交叉卓越创新中心(以下简称“中心”) / 中国科学院昆明动物研究所宿兵课题组、西藏大学欧珠罗布教授与崔超英教授课题组,联合上海营养与健康研究所/中心成员徐书华教授课题组、青海省高原医学科学研究院吴天一教授课题组等经过两年多联合攻关完成的藏族人群高原适应研究的又一阶段性成果,旨在利用长片段基因组数据从头组装藏族人群的高质量参考基因组,以期解析藏族人群基因组中的大片段结构变异对高原低氧环境的遗传贡献。
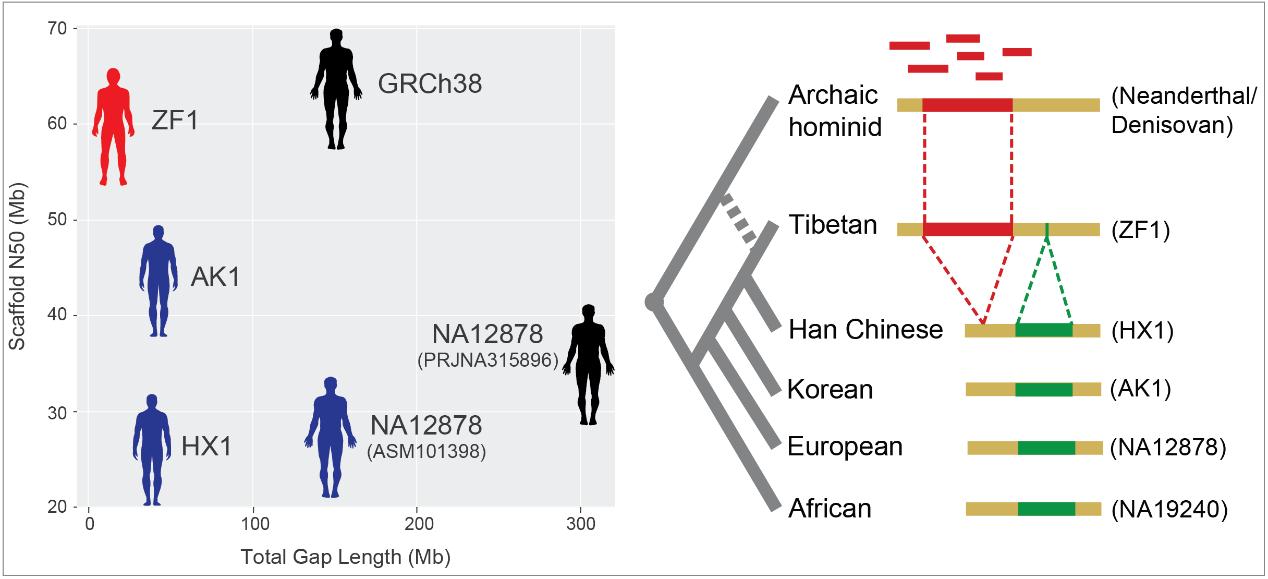
世居高原的藏族人群对极端低氧环境的适应是人类适应性进化的典型例子,一直以来受到广泛的关注。以往对于藏族高原适应的遗传分析主要集中在基于二代短读长测序数据的单核苷酸变异位点(Single Nucleotide Variants, SNVs)的研究,且发现了两个与藏族高原适应相关的关键基因EPAS1和EGLN1,解释了藏族人群较低血红蛋白浓度这一适应表型。然而,除了血红蛋白浓度,藏族其他的高原适应特征(比如较高的通气量、较低的肺动脉压等)还不能被SNVs解释。众所周知,基因组上的大尺度结构变异(Structural Variants,SVs)可能影响染色质空间结构及基因的表达调控,与疾病和进化表型也可能相关,但藏族人群基因组中的SVs对高原适应是否有贡献仅有EPAS1基因下游一个大片段缺失的一例报道,缺乏对全基因组水平SVs的系统研究。 为了系统解析藏族人群全基因组SVs,中心/中科院昆明动物所宿兵教授联合上海营养与健康研究所/中心徐书华教授、西藏大学欧珠罗布教授和青海省高原医学科学研究院吴天一院士等合作,利用三代长读长测序技术以及多种辅助组装技术,从头组装了一个高质量的藏族人参考基因组(珠峰1号,ZF1)。相比于目前已有的人类参考基因组,ZF1具有更好的序列连续性和完整性。利用该基因组,研究人员找到了17,900个ZF1中发生的 SVs,其中6,505个是ZF1有别于其他两个东亚人(HX1和AK1)的SVs。功能富集分析发现这些ZF1特有的SVs相关基因的功能显著富集在一个重要的低氧通路——GTPase活性调控通路上。通过进一步的群体分析,研究人员发现了一个发生在MKL1基因内含子上的163bp缺失,这个缺失在藏族和汉族群体中表现出显著的频率差异,且该缺失与藏族较低的肺动脉压显著相关。另外,研究人员系统评估了藏族基因组中与古人类(尼安德特人和丹尼索瓦人)共享的基因片段,发现ZF1相比于其他东亚个体的基因组有更高的共享片段比例(1.32%-1.53%)。其中一个典型的例子是发生在SCUBE2基因内含子上一个662bp的插入,分析发现该插入在藏族中富集并与藏族的肺功能显著相关。该基因组是第一个利用长片段序列从头组装的藏族人群的高质量参考基因组,并利用该基因组系统解析了藏族人群全基因组水平的结构变异元件数据集,将会为今后藏族高原适应的医学和进化研究提供重要的基础数据资源。西藏大学欧珠罗布教授、中科院昆明动物所和耀喜副研究员、中科院营养与健康研究所(马普计算生物学所)楼海一博士、邓恋博士、以及西藏大学崔超英教授为论文的共同第一作者,中心/中科院昆明动物所宿兵研究员、祁学斌副研究员和中心/中科院营养与健康研究所(马普计算生物学所)徐书华研究员为论文的共同通讯作者。 原文链接:https://academic.oup.com/nsr/advance-article/doi/10.1093/nsr/nwz160/5602645?searchresult=1 |