

高原低氧环境适应的分子机制是进化和遗传领域的重要科学问题,长期以来受到广泛的关注。近十年来,中科院昆明动物所宿兵课题组与西藏大学崔超英教授合作,通过大规模的高原藏族人群样本和比较基因组学数据分析,发现了包括EPAS1和EGLN1在内的一系列与藏族高原适应相关的候选基因(Peng et al. Mol Biol Evol 2011; Xiang et al. Mol Biol Evol 2013; Yang, et al. Hum Mut 2016; Zheng et al. Zool Res 2017; Guo et al. Zoo Res 2017; Ouzhuluobu et al. Nat Sci Rev 2020),进一步利用功能组学分析证实这些基因参与了藏族人群的高原适应(Peng et al. Mol Biol Evol 2017; He et al. Nat Sci Rev 2018)。然而,鉴于已发现的绝大部分在藏族人群中富集的适应性突变都发生在非编码区域,为系统破译这些位点的分子调控机制和功能效应带来了极大挑战。
近年来,多组学技术尤其是表观染色质状态和三维结构测序技术的发展和广泛应用,对解析这些非编码区域变异的调控机制带来了契机。为了系统解析藏族人群适应高原低氧的分子调控机制,中科院昆明动物所宿兵团队与中科院数学与系统研究院王勇团队、以及斯坦福大学和西藏大学合作,设计实验采集、测量、比较藏族适应型和汉族野生型两种脐带内皮细胞(HUVEC)在低氧和常氧条件下以及不同时间节点上的多组学数据,包括基因组、转录组、染色质可及性(ATAC-seq)和染色质空间构象(Hi-C)数据,并进一步发展了一种新的方法论框架:vPECA (Variants interpretation model by Paired Expression and Chromatin Accessibility data),整合基因组-表观组-转录组-表型层面的数据,构建以受选择调控元件为核心的基因调控网络模型,对藏族高原低氧适应的调控机制进行了系统地分析。该模型可以检测位于非编码区变异位点是否在群体中受到选择,同时又位于具有调控活性的调控元件上,并在调控网络中影响下游基因表达水平。利用该模型研究团队揭示EPAS1基因表达由受选择和不受选择两类调控元件组合调控,发现了3个位于增强子区域的功能位点,这3个位点通过削弱所在区域的染色质开放程度(可及性),进而下调EPAS1的表达,从而避免藏族人群在高原低氧环境过高表达EPAS1带来的负效应(如红细胞过度增生)。同时,研究人员还构建了EPAS1基因的下游调控网络,解析了下游受选择靶基因的调控机制,在网络中探索了高原适应相关表型的联系。
该研究提供了一个群体遗传数据结合特定细胞环境多组学数据的研究范例,包括基于多组学数据系统破译非编码区变异位点的调控方式和基于组学数据动态变化特征构建基因调控网络的统计模型,为今后多组学整合数据分析提供了一个非常有效的工具。同时,该研究进一步揭示了EPAS1对藏族低氧适应的贡献,并系统解析了低氧适应的调控网络,为今后高原适应的人群研究和低氧通路的代谢研究都提供了重要的数据。
该研究以“Chromatin accessibility landscape and regulatory network of high-altitude hypoxia adaptation”为题,于2020年10月1日在Nature Communications发表。中科院昆明动物所宿兵研究员、数学与系统研究院王勇研究员和斯坦福大学Wing Hung Wong教授为论文的共同通讯作者。信晶雪博士、张慧助理研究员,和耀喜副研究员、都仁扎那博士和白彩娟博士为论文的共同第一作者。该研究受到中科院战略先导专项、中科院动物进化与遗传卓越中心、国家自然科学基金委重点项目、国家自然科学基金委创新群体项目、中科院青促会项目、上海市科技重大专项、云南省自然科学基金的资助。
论文链接:https://www.nature.com/articles/s41467-020-18638-8
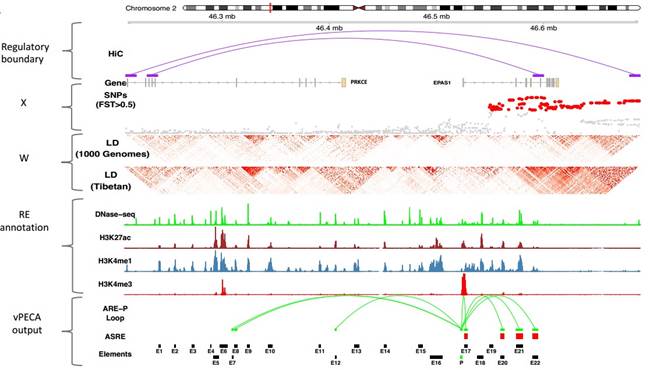
图注:利用vPECA新模型揭示EPAS1低氧适应的分子调控机制
