

高等真核生物基因组存在复杂的三维空间结构,在不同尺度下形成如染色质环(Chromatin loops)、拓扑关联结构域(TADs)、活性/非活性染色质区室(A/B compartments)和染色体域(Chromosome territories)。这些结构对于基因组稳定性的维持、基因表达的精准调控具有重要作用,从而影响细胞命运决定和表型建立[1]。经典的基因组三维结构主要是通过染色体构象捕获(3C)及其衍生方法如4Cs、5C、Hi-C,以及ChIA-PET为代表的多种形式的高通量技术揭示的。这些技术可以捕获细胞核内空间相邻的成对DNA序列[2],但无法捕获细胞群体中基因组内协同的多位点相互作用(multi-way contact)和单分子拓扑结构(single-allele topology)[3]。此外,基因组3D结构在细胞周期、发育和分化过程中动态变化,并与多个基因及调控区间的染色质相互作用相关[4]。获得细胞群体中的染色体单分子拓扑结构对于充分理解基因组的动态折叠机制和与基因调控功能的关联性至关重要。
近年来,多个实验室建立了如 ChIA-drop[5]、split-pool recognition of interactions by tag extension (SPRITE)[6]、Tri-C、multi-contact 4C[7]和Pore-C[8]等方法,用于研究染色质多位点协同相互作用和群体细胞的染色体单分子拓扑结构的捕获。这些方法中,Pore-C具有技术简单,可以同步捕获全基因组高阶多位点互作信息和DNA甲基化修饰的优点。
2023年3月6日,中国科学院昆明动物研究所侯春晖团队与中山大学中山眼科中心肖传乐团队合作在Nature Communications 在线发表了题为 High-throughput Pore-C reveals the single-allele topology and cell type-specificity of 3D genome folding的研究论文, 该工作优化建立了一种高通量的Pore-C方法,显著增加了高阶染色质互作的检测通量,并揭示了三维基因组的单分子拓扑结构多样性和细胞特异性。

在本项研究中,作者发现Pore-C技术测序通量相对较低的原因可能是与DNA交联的蛋白质没有被完全去除,导致了测序纳米孔芯堵塞。为了解决这个问题,作者优化了酶解条件,测试了多次蛋白酶解和使用混合蛋白酶的策略,大幅提高了测序产量约80%,近乎成倍降低了该技术的使用成本。
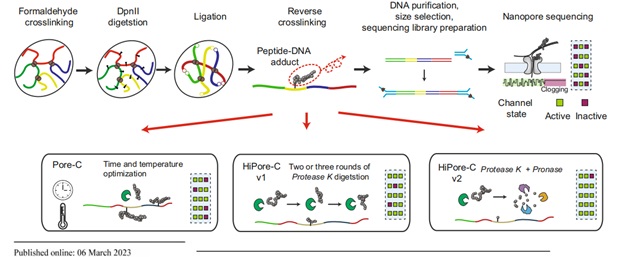
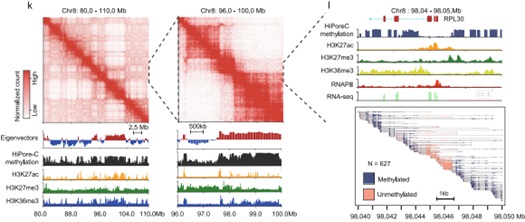
此外,作者通过整合NGMLR和Minimap2比对算法开发了MapPore-C比对流程,显著改善了比对准确性和数据利用率低的问题。作者还通过与Hi-C数据比较验证了HiPore-C能够高度重现基于Hi-C捕获的染色质环、拓扑相关结构域和染色质区室等基因组3D结构。
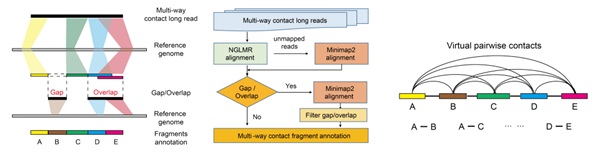
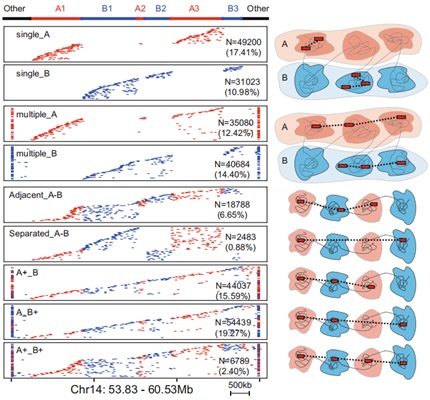
作者接着研究了染色体间高阶互作,发现大多数互作并非发生在端粒和中心粒之间,而是发生在基因组区域,并且形成两个转录活性不同的互作枢纽,其中一个枢纽基因密度、增强子密度和活跃状态染色质相关的表观遗传修饰水平都更高。作者还发现多个染色体的tRNA基因富集区域之间发生跨染色体的高频相互作用。HiPore-C高阶互作不仅发生在TAD和compartment内部,而且能够跨越多个区室、拓扑相关域和染色质环;基于直接和间接的DNA片段间相互作用构建的染色质互作图谱与常规Hi-C图谱总体相似,但是间接DNA片段互作更加倾向跨越多个结构单元。研究揭示了跨染色质结构域互作存在的广泛性,并且突出了HiPore-C技术在单分子水平解析基因组三维高阶互作的优势和重要性。
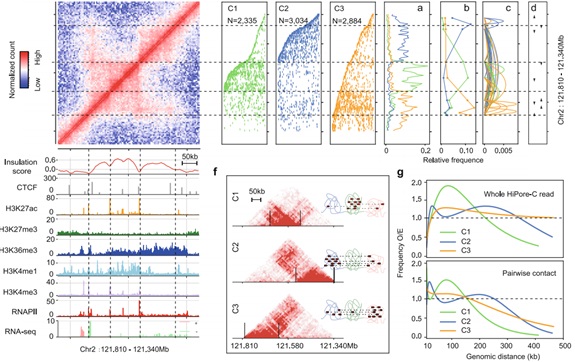
作者通过分层聚类的方法,讨论了不同类型细胞的拓扑结构中呈现的单分子拓扑结构集群,这些结构集群是类亚TAD(subTAD-like)结构域形成的基础,往往具有明显的细胞特异性。这表明了单分子拓扑结构多样性是细胞群体TAD结构域划分的基础,对我们理解基因组空间结构组织和细胞特异的基因表达间的关系具有重要意义。
此外,作者使用HiPore-C数据比较了红系K562和淋巴系GM12878细胞中在β-globin locus的高阶互作,结果发现,人ε-和γ-珠蛋白基因启动子和多个增强子之间形成了多位点同时互作、细胞特异的增强子-启动子中心,这种相互作用很可能是动态的。
最后,作者分析了HiPore-C同时捕获染色质高阶互作和DNA甲基化状态的能力,发现DNA甲基化信号与染色质环锚点间相互作用强度呈正相关,此外还可以根据DNA甲基化水平准确地区分染色质区室的类型(A vs B)。
综上所述,作者建立了HiPore-C技术,能够以前所未有的深度全面描述单分子拓扑结构的多样性,揭示了单分子拓扑结构的动态折叠比以前想象的更复杂,进一步促进了我们对三维基因组折叠规律的理解。
文章第一作者是中山大学眼科中心钟嘉泳博士和中国科学院昆明动物研究所访问学者牛龙见博士(南方科技大学公共卫生中心)。
原文链接:https://doi.org/10.1038/s41467-023-36899-x
参考文献:
1. Oudelaar, A. & Higgs, D. The relationship between genome structure and function. Nature Genetics 22, 154-168, doi:10.1038/s41576-020-00303-x (2021).
2. Jerkovic, I. & Cavalli, G. Understanding 3D genome organization by multidisciplinary methods. Nat Rev Mol Cell Biol 22, 511-528, doi:10.1038/s41580-021-00362-w (2021).
3. Allahyar, A. et al. Enhancer hubs and loop collisions identified from single-allele topologies. Nat. Genet. 50, 1151–1160 (2018).
4. Li, G. et al. Extensive promoter-centered chromatin interactions provide a topological basis for transcription regulation. Cell 148, 84–98 (2012).
5. Zheng, M. et al. Multiplex chromatin interactions with single-molecule precision. Nature 566, 558–562 (2019).
6. Quinodoz, S. A. et al. Higher-order inter-chromosomal hubs shape 3D genome organization in the nucleus. Cell 174, 744–757.e724 (2018).
7. Oudelaar, A. M. et al. Single-allele chromatin interactions identify regulatory hubs in dynamic compartmentalized domains. Nat. Genet. 50, 1744–1751 (2018).
8. Deshpande, A. S. et al. Identifying synergistic high-order 3D chromatin conformations from genome-scale nanopore concatemer sequencing. Nat. Biotechnol. 40, 1488–1499 (2022).
