

大规模、高精度、高时效地获取物种分布动态,是有效开展生物多样性保护和管理、实现“昆明-蒙特利尔全球生物多样性框架”目标所面临的全球性挑战。传统的生物多样性监测方法往往难以兼顾覆盖范围、分类阶元广度和监测效率,这一难题在地形与生境极其复杂的山地生态系统中尤为突出。
为了应对这一全球性挑战,中国科学院昆明动物研究所生物多样性保护研究中心、云南省高黎贡山生物多样性重点实验室的Douglas Yu团队联合国内外多个合作团队,将研究聚焦于全球生物多样性保护的战略要地——高黎贡山。该地区作为全球三大生物多样性热点的唯一交汇区,是名副其实的“世界物种基因库”与我国西南生态安全的“绿色长城”。在此开展系统性研究,旨在验证环境DNA宏条形码技术能否为这一极端复杂且至关重要的区域提供一套高效、可靠的监测方案,并进而为全球生物多样性监测提供一套可推广的高效解决方案。

研究团队在高黎贡山脉横跨30,000平方公里的广阔区域内,仅用33个野外工作日和69个实验室工作日,便成功检测到389种脊椎动物。这一惊人的效率揭示了前所未有的物种多样性信息,其中包括了众多珍稀濒危物种,如极度濒危的马来穿山甲(Manis javanica)、濒危的豺(Cuon alpinus)、肖氏乌叶猴(Trachypithecus shortridgei)和中华小熊猫(Ailurus styani),以及国家一级保护动物贡山羚牛(Budorcas taxicolor)和白尾梢虹雉(Lophophorus sclateri)等。
该研究的核心突破在于开发并应用了名为“OccPlus”的eDNA专用多物种占域模型。这一创新模型能够精准区分真实的稀有物种信号与潜在的eDNA污染。以贡山羚牛为例,OccPlus能够识别出其在南部地区的微弱检测信号为假阳性,从而精准地重现了该物种仅分布于北部的真实格局,而传统占域模型则无法完成此类关键的校正(图1)。这一技术突破极大地提升了eDNA监测数据的可靠性与质量,为生成可信的物种分布格局提供了强有力的统计学保障。
基于高质量的eDNA数据,研究为高黎贡山的生物多样性保护提供了数项关键的新认知。首先,研究以空前的采样规模和数据分辨率,清晰再现了高黎贡山水系鱼类的物种丰富度由北向南递增的地理格局。在此基础上,研究进一步揭示了保护区对外来鱼类的限制作用更多源于高海拔等自然地理屏障的间接结果。尤为重要的是,通过OccPlus模型分析,研究还成功锁定了由旅游开发和水产养殖驱动的局部入侵“热点”区域(丙中洛镇),证实了人为因素是当前外来物种在该地区扩散的关键驱动力(图2)。其次,研究为高黎贡山保护区的管理成效提供了直接的、大尺度的分子生态学证据,结果清晰地表明,本土野生陆生动物在保护区内的占域率显著高于保护区外(图3, 4)。
此项研究是首个发表于生态学顶级期刊的、针对高黎贡山地区开展的大规模eDNA监测工作,它不仅是一次技术应用的成功展示,更彰显了昆明动物研究所生物多样性保护研究中心及云南省高黎贡山生物多样性重点实验室在全球生物多样性热点地区开展前沿科学研究的领先实力。通过整合环境DNA宏条形码技术与前沿的统计模型,该团队建立了一套能够高效、可靠地应用于全球复杂山地生态系统的生物多样性监测“黄金标准”,为全球的生态保护与管理实践树立了新的标杆。
研究成果以“High quality, granular, timely, trustworthy, and efficient vertebrate species distribution data across a 30,000 km2 protected area complex”为题,近日发表于《Ecology Letters》。昆明动物所季吟秋副研究员、英国埃塞克斯大学Alex Diana博士和昆明动物所李学友副研究员为本文共同第一作者,昆明动物所Douglas W. Yu研究员和美国哥伦比亚大学Viorel D. Popescu副教授为共同通讯作者。该项研究的顺利完成还得益于所内外专家的通力合作,生物多样性保护研究中心的车静研究员团队(两栖爬行类)、蒋学龙研究员团队(兽类)、陈小勇研究员团队(鱼类)以及鸟类学专家吴飞副研究员、董峰研究员等成员为研究提供了关键的物种分布背景和分类学支持。此外,英国伦敦玛丽女王大学Eleni Matechou教授和伦敦大学学院Jim E. Griffin教授共同参与了核心统计模型的构建。
该研究得到了国家重点研发计划、国家自然科学基金、云南省“兴滇英才”支持计划等项目的资助。
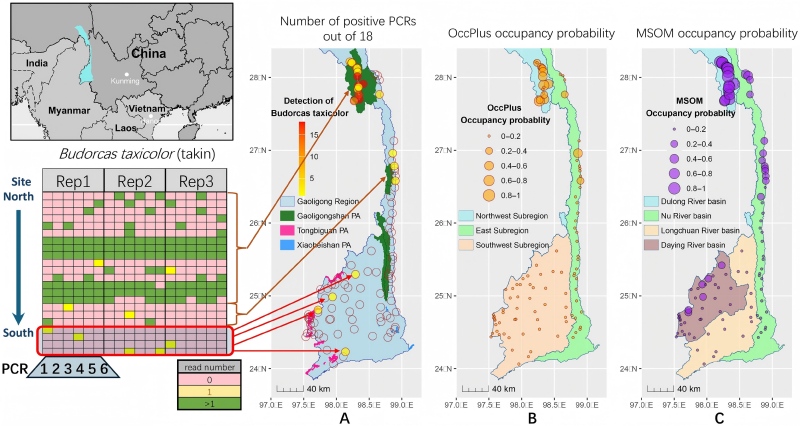
图1 OccPlus模型与原始检测数据、传统多物种占域模型(MSOM)在校正eDNA数据错误上的对比(以贡山羚牛为例)。左图表格展示了样点中检测到贡山羚牛的原始PCR数据,颜色深浅代表DNA序列数量(图中A)。OccPlus模型的先进算法能够识别出高黎贡山南部的4个弱信号检测(如18次PCR仅1次阳性)为假阳性,从而精准地重现了贡山羚牛仅分布于北部的真实格局(图中B)。相比之下,传统的MSOM模型则无法有效校正此类错误(图中C)。
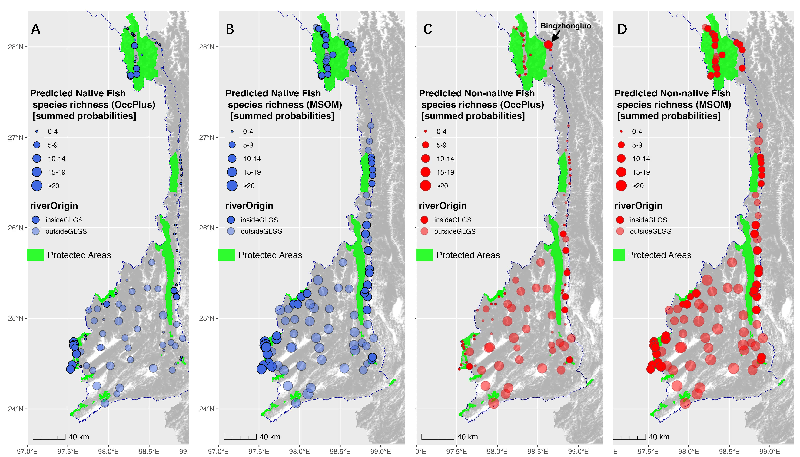
图2 本土与外来鱼类物种丰富度的空间格局。该图对比了OccPlus(A, C)与MSOM(B, D)两种模型对本土鱼类(蓝色)和外来鱼类(红色)物种丰富度的预测结果。OccPlus模型更清晰地揭示了鱼类多样性由北向南递增的地理梯度,并精准锁定了由人类活动驱动的局部入侵“热点”—丙中洛。
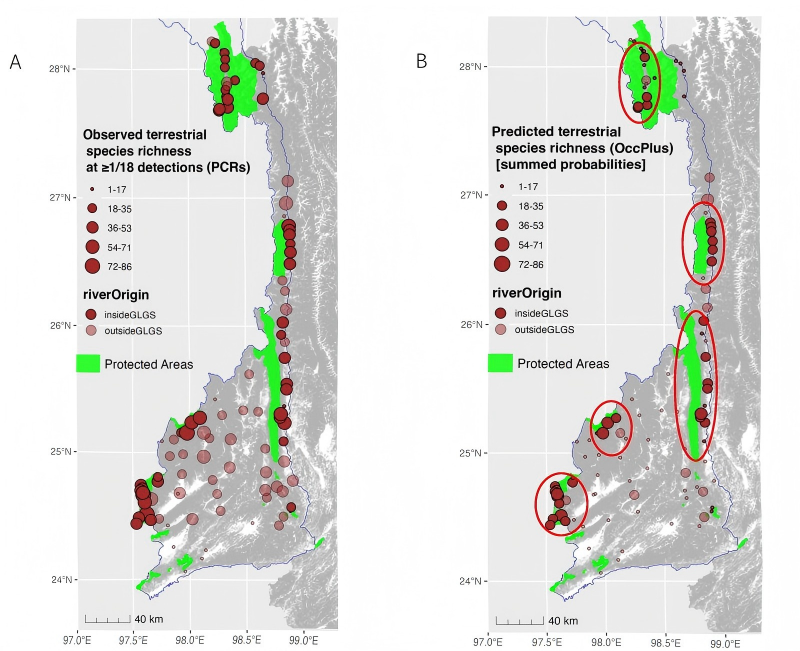
图3 陆生脊椎动物物种丰富度的观测值与占据模型校正值的对比。(A)各样点陆生脊椎动物的原始检测物种数;(B)基于OccPlus占据模型的校正结果,通过各物种出现概率累加计算的occupancy-corrected物种丰富度。B图中的红色圆圈表示圈住的样点水流来源于保护区内。
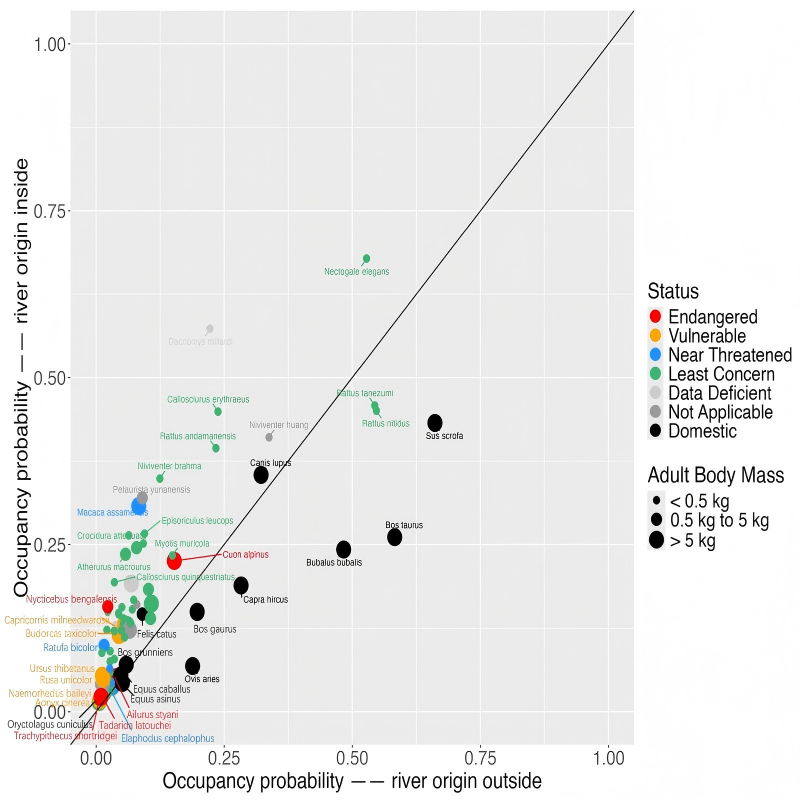
图4 基于OccPlus获得的兽类物种在保护区内外出现概率的对比。每个点代表一个物种,点的颜色代表其IUCN红色名录等级(黑色为家养物种),点的大小代表了物种的成年个体平均体重,X轴是物种在保护区外的出现概率,Y轴是物种在保护区内的出现概率。该图直观地展示了绝大多数本土野生兽类在保护区内的出现概率更高,而家养动物则相反,为保护区的有效性提供了直接证据。
